Alvaro Mallagarays Forschungsgruppe
Unsere Forschung fokussiert sich auf die Entwicklung und Anwendung NMR-basierter Methoden zur molekularen Charakterisierung biologischer Systeme. Im Mittelpunkt stehen metabolische und glykosylierungsbezogene Signaturen in biologischen Flüssigkeiten sowie deren Veränderungen in entzündlichen, onkologischen und infektiösen Erkrankungen mit dem Ziel der Biomarkeridentifikation. Ein weiterer Schwerpunkt liegt auf der strukturellen Glykobiologie und Virologie. Wir untersuchen die molekularen Mechanismen der Glykankennung durch Viren wie Noroviren und Coronaviren sowie die Verarbeitung komplexer Polysaccharide und N-Glykane im menschlichen Darmmikrobiom. Durch die Kombination experimenteller NMR-Spektroskopie mit computergestützter Datenanalyse und interdisziplinären Kooperationen verfolgen wir einen translationalen Ansatz von der Grundlagenforschung bis zur klinischen Anwendung.
Bisherige und aktuelle Forschung
Projekt 1: Schnelle Quantifizierung von plasmaassoziierten N-Glykosylierungsprofilen bei Krebsprogression und Entzündungsprozessen mittels NMR
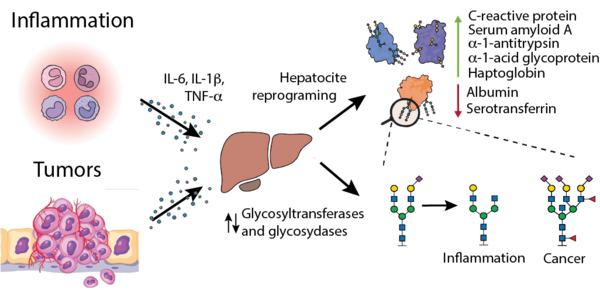
Die Proteinglykosylierung ist die häufigste posttranslationale Modifikation in menschlichen Zellen und führt zu einer großen Vielfalt an Glykokonjugaten. Abnorme Glykosylierungsmuster zirkulierender Proteine werden aufgrund neoplasiebedingter hepatischer Reprogrammierung als starke diagnostische Marker für Krebserkrankungen genutzt und sind auch bei zahlreichen entzündungsassoziierten Erkrankungen beschrieben.
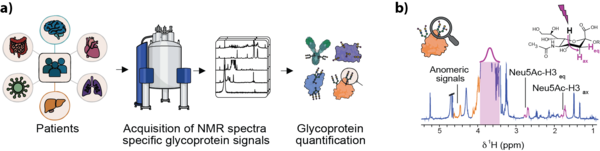
In diesem Projekt entwickeln wir schnelle, quantitative NMR-Methoden zur Charakterisierung entzündungsassoziierter Glykoproteinprofile direkt in nativem humanem Serum und Plasma. Ziel ist es, glykosylierungsbasierte Biomarker in skalierbare klinische Diagnostikansätze und groß angelegte Kohortenstudien zu überführen. Der Fokus liegt auf der Extraktion protein- und glykosylierungsspezifischer Informationen aus standardisierten 1D-NMR-Spektren bei minimaler Probenhandhabung sowie auf robusten, automatisierten und instrumenten- sowie standortübergreifend reproduzierbaren Workflows.
Neben Signalen niedermolekularer Metabolite und Lipoproteine enthalten Blut-NMR-Spektren charakteristische Resonanzen protein-gebundener Glykane, wie GlycA und GlycB. Darüber hinaus weisen Blut-NMR-Spektren eine Vielzahl glykanabgeleiteter Signale auf, die Informationen über die Glykankomposition liefern und stark auf entzündliche und onkogene Prozesse reagieren. Unsere Arbeiten zielen darauf ab, neue Experimente und informatische Werkzeuge zu entwickeln, um diese bislang weitgehend unerschlossenen NMR-Signale zu extrahieren und mechanistische Interpretationen von Veränderungen der Glykoproteinkonzentrationen sowie klinisch relevante Glykoprofile zu ermöglichen.
Die Entwicklung neuer Biomarker wird durch enge Kooperationen mit klinischen Partnern und die systematische Translation in unterschiedliche Kohortenstudien vorangetrieben. Mithilfe unserer patentierten NMR-basierten Technologie zur Zuordnung und Quantifizierung glykoproteinspezifischer Beiträge und Glykosylierungsmuster in Serum und Plasma streben wir die Identifikation und Validierung von Biomarkerprofilen für ein breites Spektrum entzündlicher und onkogener Pathologien an. Dazu zählen u. a. chronisch-entzündliche Darmerkrankungen, die Parkinson-Erkrankung, systemische Sklerose, MASLD und deren Progression bis zum hepatozellulären Karzinom sowie weitere Krebserkrankungen wie kolorektales Karzinom, Cholangiokarzinom, uveales Melanom und multiples Myelom.
Projekt 2: Kontrolle des komplexen Polysaccharidstoffwechsels im menschlichen Darmmikrobiom
Das Darmmikrobiom spielt eine zentrale Rolle für die menschliche Gesundheit, indem es die Verdauung unterstützt, das Immunsystem formt und vor Pathogenen schützt. Zahlreiche Erkrankungen stehen mit Störungen dieser mikrobiellen Gemeinschaften ("Dysbiose") in Verbindung. Ein wesentlicher Faktor für die Zusammensetzung des Mikrobioms ist das kontinuierliche Angebot komplexer Kohlenhydrate aus der Nahrung und der mukösen Darmschleimhaut. Da diese Zucker eine zentrale Energiequelle darstellen, haben Darmbakterien hochspezialisierte Mechanismen zu deren Abbau, Bindung und Aufnahme entwickelt, deren molekulare Grundlagen jedoch noch unvollständig verstanden sind.
Ziel dieses Projekts ist es, auf molekularer Ebene zu verstehen, wie Darmbakterien komplexe Polysaccharide und N-Glykane verarbeiten. Bestimmte Darmbakterien, insbesondere Bacteroidetes, organisieren die dafür notwendigen Gene in kompakten "Werkzeugkästen", sogenannten Polysaccharid-Utilisierungs-Loci (PULs). Diese kodieren Enzyme zum gezielten Abbau spezifischer Kohlenhydrate sowie Proteine zur Bindung und zum Transport der Abbauprodukte in die Zelle. Das Verständnis dieser Prozesse eröffnet neue therapeutische Strategien zur gezielten Modulation des Mikrobioms, etwa zur Förderung nützlicher Bakterien oder zur Eindämmung entzündungsfördernder oder antibiotikaresistenter Stämme.
Wir kombinieren NMR-Spektroskopie mit komplementären strukturbiologischen Methoden (Massenspektrometrie, Kryo-EM, Kristallographie u. a.), um die Funktionsweise zentraler Enzyme, deren Substraterkennung sowie das Zusammenspiel verschiedener Proteine bei der Aufnahme von Zuckern in bakterielle Zellen zu entschlüsseln. Durch die detaillierte Kartierung dieser Mechanismen schaffen wir die Grundlage für präzise Interventionen, die den mikrobiellen Stoffwechsel – und damit die Zusammensetzung des Mikrobioms – gezielt und medizinisch nutzbar verändern können.
Projekt 3: Untersuchung der Interaktion von Noroviren mit zellulären Anheftungsfaktoren für die Infektion
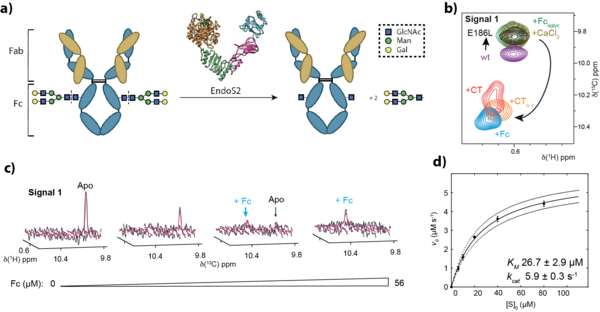
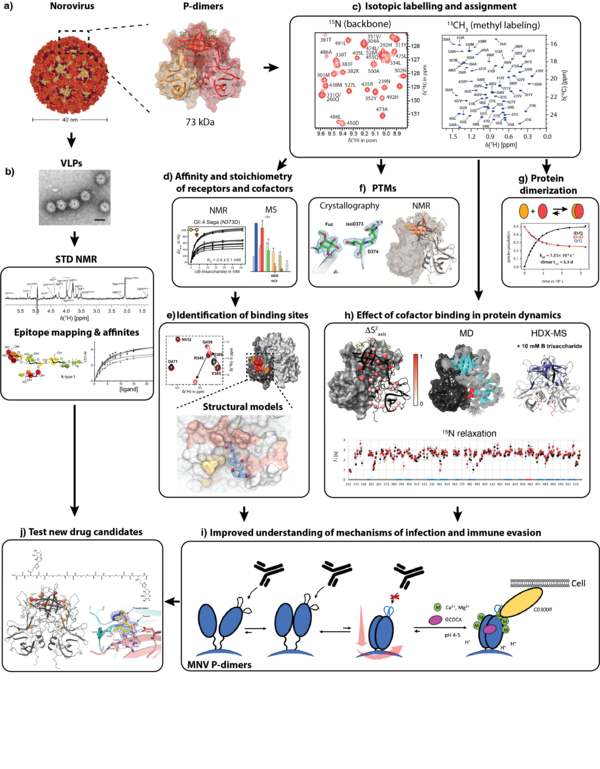
Noroviren sind eine der häufigsten Ursachen akuter Gastroenteritiden. Der Infektionsprozess beginnt mit der Bindung des viralen Kapsids an Histo-Blutgruppenantigene (HBGAs) der gastrointestinalen Mukosa. In diesem integrativen Projekt nutzen wir NMR-basierte strukturelle Glykobiologie, um zu untersuchen, wie humane und murine Norovirus-Kapsidproteine Wirtsglykane und gallensäurebasierte Kofaktoren erkennen, die für die Infektion erforderlich sind. Ziel ist die Entwicklung hochselektiver Eintrittsinhibitoren gegen Noroviren.
Der methodische Kern kombiniert liganden- und proteinbeobachtende NMR-Ansätze. Saturation-Transfer-Difference-(STD-)NMR-Titrationen ermöglichen Epitop-Mapping und Bindungsanalysen sowohl an P-Dimeren als auch an assemblierten virusähnlichen Partikeln (VLPs). 1H,15N-TROSY-HSQC-Titrationen an vollständig 2H,15N-markierten P-Domänen liefern einen direkten und stringenten Bindungsnachweis unter nahezu physiologischen Bedingungen. Zur Untersuchung größerer Systeme und subtiler allosterischer Effekte wurden Methyl-TROSY-Experimente an selektiv methylmarkierten Kapsiddomänen etabliert und durch orthogonale Validierung mittels Massenspektrometrie, Röntgenkristallographie und langzeitiger Molekulardynamiksimulationen in Zusammenarbeit mit spezialisierten Partnergruppen ergänzt.
Über mehrere humane und murine Norovirus-Stämme hinweg konnten wir zeigen, dass weit verbreitete GII.4-Kapsiddomänen HBGAs unabhängig mit niedrigen millimolaren Affinitäten binden, wobei Fucose das minimale Erkennungsmotiv darstellt und keine Bindung an nicht-fukosylierte Sialoglykane erfolgt. Darüber hinaus identifizierten wir wesentliche Ursachen für irreführende "komplexe Bindungssignale", darunter Glykan-Clusterbildung in der nativen Massenspektrometrie sowie eine zeitabhängige N373→isoAsp-Umwandlung, die die HBGA-Bindung abschwächen kann. Über Glykane hinaus zeigte die Kombination aus Backbone- und 13C-Methyl-NMR in Verbindung mit Molekulardynamik das Vorhandensein einer niedrigaffinen, flexiblen Gallensäure-Bindetasche in GII.4-Kapsiden. Insgesamt definiert dieses Projekt zentrale Aspekte der Norovirus-Ligandenerkennung neu, liefert Best-Practice-Leitlinien für reproduzierbare Bindungsstudien über verschiedene Plattformen hinweg und schafft mechanistische Grundlagen für rationale Eintrittsinhibitor-Konzepte, einschließlich multivalenter Glykomaterialien und sondengestützter Screening-Ansätze mit paramagnetischen Glykanen.